HIGHLIGHTS: Può il metodo determinare i risultati? Alcune osservazioni sul lavoro “Correlative climatic niche models predict real and virtual species distributions equally well”
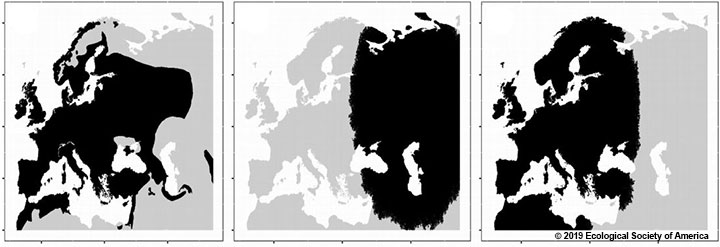
Un interessante articolo di Journé et al. (2019) è stato recentemente pubblicato sulla rivista Ecology (https://doi.org/10.1002/ecy.2912). Lo studio affronta un problema complicato e spesso dibattuto: può il clima essere utilizzato come predittore nella modellistica previsionale (Species Distribution Modelling, SDM – o, come hanno scritto nel loro articolo, Correlative Species Distribution Modeling, cSDM) – per modellizzare (e prevedere) la distribuzione delle specie? Questo argomento è frequentemente dibattuto nella letteratura del settore, e ha spesso costretto i ricercatori a individuare l’insieme di variabili climatiche più rappresentativo per ogni modello onde evitare problemi di collinearità ma includendo, al tempo stesso, quante più informazioni possibili nel calcolo. L’articolo segue rigorosamente tutte le linee guida principali per la modellizzazione: (i) utilizza i dati sulla presenza delle specie per modellare i taxa analizzati tra cui 132 specie terrestri europee, scelte a caso all’interno di cinque gruppi tassonomici (tre gruppi di vertebrati e due gruppi di piante” (!); ( ii) un’analisi delle componenti principali è stata utilizzata per trasformare i predittori ottenendo un insieme (minore) di dati non collineari ma che includono al tempo stesso tutta la variabilità ambientale (Marchi & Ducci, 2018); (iii) il pacchetto biomod2 (Thuiller et al. 2016) del linguaggio R (R CoreTeam 2018) è stato selezionato come ambiente di lavoro; (iv) GAM e MAXENT sono stati usati come algoritmi di modellazione, tra i più utilizzati e performanti in letteratura insieme al Random Forest (Pecchi et al., 2019); (v) pseudo assenze e valori AUC e TSS (ovvero valori derivanti da cross validazione) sono stati calcolati ed applicati correttamente (Barbet-Massin et al., 2012).
Tuttavia, vale la pena di discutere dell’approccio basato sul “diffusion model” che gli autori hanno usato per creare 1.320 specie virtuali la cui distribuzione era causalmente completamente indipendente dal clima (a detta loro). Traducendo quanto riportato nel testo, l’uso di una “colonizzazione spaziale graduale di pixel attorno a un punto iniziale” (anche se casuale) “fino al raggiungimento della dimensione finale (ovvero, la stessa dimensione dell’intervallo delle specie reali corrispondenti)” non garantisce che tale distribuzione spaziale delle specie virtuali modellizzate possa essere completamente indipendente dal clima. Al contrario, è ben noto che più due località sono vicine geograficamente, più è probabile che il loro clima sia simile. Il clima infatti è uno dei fenomeni spazialmente più autocorrelati che possiamo osservare in natura. Non a caso i parametri climatici (temperature, precipitazioni) sono spesso interpolati con il metodo kriging, che è il metodo che sfrutta l’autocorrelazione per eccellenza. Per questo motivo, a mio avviso, sarebbero necessarie ulteriori analisi, spazialmente indipendenti per verificare se il clima possa essere usato con successo (e affidabilità) in SDM oppure no. Secondo il mio parere e sulla base dei risultati dello studio citato, la risposta alla domanda se il clima sia un buon predittore della distribuzione delle specie è facile: la risposta è ovviamente sì, e dipende da come le unità campionarie simulate (cioè i dati di presenza) sono distribuite spazialmente.
Are the data driving the results? Some observations on the paper “Correlative climatic niche models predict real and virtual species distributions equally well”
An interesting paper by Journé et al. (2019) has been recently published in Ecology Journal (https://doi.org/10.1002/ecy.2912). The study deals with a complicated and annoying issue: can climate be successfully used as predictor in Species Distribution Modelling techniques (SDM) – or, as they wrote in their paper, Correlative Species Distribution Models (cSDMs) – to predict (and forecast) species distribution properly? This topic is often debated in literature, forcing researcher to find the most representative set of climatic variables to avoid collinearity and include as much information as possible in the computation. The paper strictly follows all the main guidelines for modelling: (i) species presence data are used to model analysed taxa including “132 European terrestrial species, chosen randomly within five taxonomic groups (three vertebrate groups and two plant group” (!); (ii) PCA was used to transform predictors obtaining a set of (less) non collinear predictors (Marchi & Ducci 2018); (iii) the biomod2 package (Thuiller et al. 2016) of R language (R CoreTeam, 2018) was selected as modelling framework; (iv) GAM and MAXENT were used as modelling algorithms, among the most used and well-referenced in literature with Random Forest (Pecchi et al. 2019); (v) pseudo absences and AUC and TSS values (i.e., cross validation) were applied properly (Barbet-Massin et al. 2012).
Nevertheless, the “diffusion model approach” the Authors used to create the 1,320 virtual species “whose distribution was causally fully independent from climate“, is worth to be discussed. The use of a “gradual spatial colonization of pixels around a starting point (even if random) until the final range size was reached (i.e., the same range size as the corresponding real species)” does not ensure the spatial distribution of the virtual species to be really unlinked with climate. On the contrary, the closer two locations are, the more similar the climate is. Climate is one of the most spatially-autocorrelated phenomena we can observe in nature, and climatic parameters are often interpolated by kriging, which is the autocorrelation method for excellence. For this reason, further spatially-independent analyses are needed to test whether climate could be successfully (and reliably) used in SDM. In my opinion, according to the results of the mentioned study, the answer to the question whether climate is a good predictor of species distribution is easy: yes, it depends on how simulated sample units (i.e., occurrence data) are spatially distributed.
Bibliografia/References
- Barbet-Massin, M., Jiguet, F., Albert, C.H., Thuiller, W., 2012. Selecting pseudo-absences for species distribution models: How, where and how many? Methods Ecol. Evol. 3: 327-338. – doi :https://doi.org/10.1111/j.2041-210X.2011.00172.x
- Marchi, M., Ducci, F., 2018. Some refinements on species distribution models using tree-level national forest inventories for supporting forest management and marginal forest population detection. iForest – Biogeosciences and Forestry 11: 291–299. – doi: https://doi.org/10.3832/ifor2441-011
- Pecchi, M., Marchi, M., Burton, V., Giannetti, F., Moriondo, M., Bernetti, I., Bindi, M., Chirici, G., 2019. Species distribution modelling to support forest management. A literature review. Ecol. Modell. 411, 108817. – doi: https://doi.org/10.1016/j.ecolmodel.2019.108817
- R CoreTeam, 2018. R: A language and environment for statistical computing. https://www.r-project.org/
- Thuiller, W., Georges, D., Engler, R., 2016. biomod2: Ensemble platform for species distribution modeling. Ecography 32 (3): 369-373. – doi: https://doi.org/10.1111/j.1600-0587.2008.05742.x
Info Autori
Istituto di Bioscienze e Biorisorse (IBBR)
Consiglio Nazionale delle Ricerche (CNR)





